+400-6900-195
公司新闻
RNAi指南
|
RNAi技术发展的历程 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
1998:植物基因中基因沉默现象的发现 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
RNAi实验原理 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
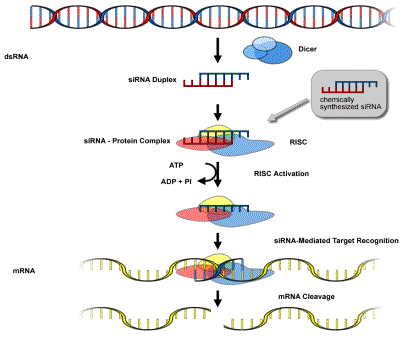
RNA干扰(RNA interfering,RNAi)现象是由与靶基因序列同源的双链RNA(double-stranded RNA,dsRNA)引发的广泛存在于生物体内的序列特异性基因转录后的沉默过程。细胞中的核糖核酸酶III家族成员之一的,dsRNA特异性的核酸酶Dicer将dsRNA裂解成由21-25个核苷酸组成的小干扰RNA (small interfering RNA,siRNA),随后siRNA作为介导子引起特异性地降解相同序列的mRNA,从而阻断相应基因表达的转录后基因沉默机制。 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
研究基因功能的新工具 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
由于RNAi具有高度的序列专一性和有效的干扰,可以特异地将特定的基因沉默,从而获得基因功能丧失或基因表达量的降低,因此可以作为功能基因组学的一种强有力的研究工具。目前已经进入应用RNAi研究基因功能的告诉发展的时期,抓住这个强力有力的工具,抓住机遇,加速基因功能的研究。 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
基因治疗 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
随着对疾病本质的深入了解和新的分子生物学技术的出现,广义上把凡是通过改变人体内的基因表达以达到治疗疾病的方法均可称为基因治疗(gene therapy)。用RNAi特异性地抑制如艾滋病病毒基因、肝炎病毒基因、癌基因、癌相关基因或突变基因的过度表达,使这类基因保持在静默或休眠状态,从而有望用这种新的手段治疗各种病毒性疾病和恶性肿瘤等疑难病症。 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
药物开发 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
RNAi技术可以作为寻找新的药物靶标的工具,可以快速、大规模、高通量的对发现的药物靶基因进行功能分析。同时,siRNA基因药物具有高度的序列特异性和非常小的毒副作用。利用这个技术可以用于确定人类细胞中的疾病途径,为小分子药物筛选靶物质,以及建立新的生物制药方法和诊断学方法。再者,它还能帮助鉴定药物作用的生化模式,以及其他与此作用相关的基因。RNAi技术已被作为高通量药物靶标识别和确认的工具之一。 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
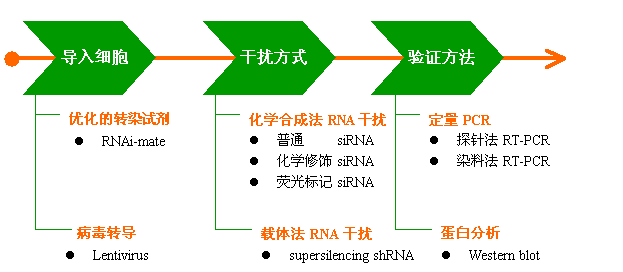
吉玛公司为客户提供RNA干扰技术品种齐全的产品 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
RNAi方法的选择 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||

|
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
化学合成法的优越性 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
GenePharma公司可以根据用户要求提供高质量的化学合成siRNA。l 最省时—四天内到货l 最省事—设计都可以委托GenePharma公司免费完成, 收到siRNA oligo后直接与转染试剂混合就可开始实验l 最省钱—500元/对, 三对里就应该有一对抑制效率在70%以上 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
化学修饰stableTM siRNA oligo |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
RNAi技术的最大难题就是化学合成siRNA稳定性问题。GenePharma化学修饰stableTM siRNA最大的目标就是一次性获得最佳的实验数据。我们的方法就是使用RNAi-Mate转染试剂将化学修饰stableTM siRNA导入哺乳动物细胞中,获得如下满意结果:
更长的作用时间 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
化学修饰stableTM siRNA oligo与普通的siRNA oligo性能对照表 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
shRNA表达载体法 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
多数的shRNA表达载体依赖三种RNA聚合酶III 启动子(pol III)中的一种,操纵一段小的发夹RNA(short hairpin RNA, shRNA)在哺乳动物细胞中的表达。这三类启动子包括大家熟悉的人源和鼠源的U6启动子和人源的H1启动子。之所以采用RNA pol III启动子是由于它可以在哺乳动物细胞中表达更多的小分子RNA,而且它是通过添加一串(3到6个)U来终止转录的。要使用这类载体,需要订购2段编码短发夹RNA序列的DNA单链,退火,克隆到相应载体的pol III 启动子下游。GenePharma公司的shRNA表达载体的优点在于可以进行较长期研究,载体可以在细胞中持续抑制靶基因的表达,持续数星期甚至更久。 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
siRNA 套餐服务 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
客户只需提供基因序列GeneID或者Accession number, 我们免费帮您设计选择合适的位点,保证至少有一对可以有效的抑制相应基因的表达,抑制效率可达70%以上,如果没有筛选出有效的siRNA 片段,吉玛公司为您免费再次设计与合成。 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
shRNA套餐服务 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
shRNA表达载体带有抗生素标记,可以在细胞中持续抑制靶基因的表达,持续数星期甚至更久。对于一个已知有效的 siRNA序列(例如,经过siRNA合成方法筛选获得的),需要维持较长时间的基因沉默时,推荐使用shRNA载体系统。GenePharma公司致力于提供最先进和最方便的shRNA相关工具,最近推出新一代shRNA表达质粒,一种高效即用型载体,该载体在细胞内可以持续产生shRNAs,从而达到持久抑制目标基因表达的目的。针对目的基因设计并制备4个shRNA载体,确保4个shRNA中至少一个在mRNA水平的基因的抑制效率在70%以上. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
RNAi经典参考文献 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
1.Cogoni C, and Macino G. (2000) Post-transcriptional gene silencing across kingdoms. Genes Dev 10: 638-643. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
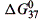 的值
的值